南湖新闻网讯(通讯员 谢胜松,陶大刚)近日,赵书红教授团队建立了基于CRISPR/Cas12系统介导的核酸裸眼可视化检测新技术,结合卷积神经网络算法开发了一个能提高检测灵敏度和智能化结果判读的手机APP软件,可方便实现现场化高灵敏高特异核酸分子诊断。
核酸检测已广泛应用于许多领域,如基因分型、食品安全检测、肉源成分鉴定、转基因成分和病原微生物检测等。尽管目前基于聚合酶链反应(PCR)的核酸检测技术灵敏度高,但这些方法需要昂贵的设备、训练有素的技术人员和干净的实验室环境,因此限制了它们在发展中国家和缺乏实验设施的养殖企业使用。随后科研人员开发了多种等温扩增技术,主要包括重组酶聚合酶扩增(RPA)和环介导等温扩增(LAMP)等。然而,绝大多数这些基于LAMP/RPA的检测可能存在非特异性核酸扩增,这意味着难以将非预期的扩增子与正确的目标扩增子区分开来,从而导致核酸检测结果不可靠。
为了克服以上问题,研究团队将目标锁定在近年飞速发展起来的新一代基因编辑技术——CRISPR/Cas12技术。该技术主要采用具有双链DNA及单链DNA切割活性的Cas12,在引导RNA (crRNA)的帮助下,Cas12与待检测的靶标双链DNA结合后改变构象,其不仅可特异性地切割目标DNA,还可非特异性地切割周围的单链DNA。研究团队2020年在国际期刊ACS Synthetic Biology杂志发表的一项研究中发现,通过筛选6种不同荧光基团修饰的单链DNA报告基因,鉴定到能检测荧光信号的新的报告基因,由此建立了基于CRISPR/Cas12系统介导的荧光增强可视化核酸检测技术。有趣的是,在一次偶然的实验中,研究人员意外发现,其中有一种新修饰的单链DNA报告基因被Cas12切割后,可以明显观察到溶液颜色发生改变。随后,研究团队通过扩大筛选范围,对多达16种单链DNA报告基因进行系统深入研究,最终发现ROX荧光基团修饰的报告基因具有优越的裸眼检测性能,其能随着浓度的变化,在被Cas12切割后,导致反应溶液的颜色发生明显改变,由此有助于通过肉眼直接观察到核酸检测信号。
为了提升核酸检测灵敏度,以及对检测信号进行智能化与标准化判读。结合卷积神经网络算法,研究团队合作开发了一个手机APP(MagicEye),其能准确分辨单个或八连管,以及反应溶液颜色的变化。通过手机拍照,用户将检测数据发送到云端服务器,经过MagicEye软件分析后,其可在手机端快速接收到核酸检测报告。接着,为了避免核酸检测时受气溶胶胶污染造成的假阳性干扰,研究团队进一步对实验条件进行了优化,由此实现了一管法核酸快速检测,即将Cas12/crRNA置于管盖,当核酸扩增反应完成后,混合两者即可实现不开盖检测核酸分子。新方法检测速度快,从核酸提取到结果判读仅需45分钟,且只需简单的加热装置即可完成反应,特别适合用于现场即时POCT核酸检测。通过与荧光定量PCR技术相比,新方法检测准确性为100%,能实现单分子检测,且成本更低。由此研究团队建立了一种名为“RAVI-CRISPR”的快速可视化核酸检测平台,为今后用于基因分型和疾病诊断提供了新技术。
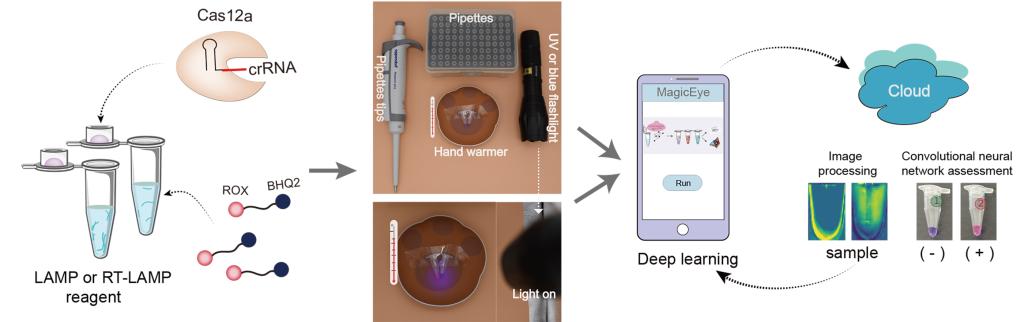
RAVI-CRISPR核酸裸眼可视化检测技术
学院副教授谢胜松、硕士研究生陶大刚和徐兵荣、博士后付玉华为该论文的共同第一作者,必赢766net手机版赵书红教授、刘小磊教授和李新云教授为论文共同通讯作者。该研究得到国家自然科学基金、国家重点研发计划项目等项目资助。
审核人:赵书红
【英文摘要】
Rapid diagnosis based on naked-eye colorimetric detection remains challenging, but it could build new capacities for molecular point-of-care testing (POCT). In this study, we evaluated the performance of 16 types of single-stranded DNA-fluorophore-quencher (ssDNA-FQ) reporters for use with clusters of regularly spaced short palindrome repeats (CRISPR)/Cas12a-based visual colorimetric assays. Among them, nine ssDNA-FQ reporters were found to be suitable for direct visual colorimetric detection, with especially very strong performance using ROX-labeled reporters. We optimized the reaction concentrations of these ssDNA-FQ reporters for a naked-eye read-out of assay results (no transducing component required for visualization). In particular, we developed a convolutional neural network algorithm to standardize and automate the analytical colorimetric assessment of images and integrated this into the MagicEye mobile phone software. A field-deployable assay platform named RApid VIsual CRISPR (RAVI-CRISPR) based on a ROX-labeled reporter with isothermal amplification and CRISPR/Cas12a targeting was established. We deployed RAVI-CRISPR in a single tube toward an instrument-less colorimetric POCT format that required only a portable rechargeable hand warmer for incubation. The RAVI-CRISPR was successfully used for the high-sensitivity detection of severe acute respiratory syndrome coronavirus 2 (SARS-CoV-2) and African swine fever virus (ASFV). Our study demonstrates this RAVI-CRISPR/MagicEye system to be suitable for distinguishing different pathogenic nucleic acid targets with high specificity and sensitivity as the simplest-to-date platform for rapid pen- or bed-side testing.
论文链接:https://pubs.acs.org/doi/full/10.1021/acssynbio.1c00474

